stLearn 分析
前言
IMPORTANT
stLearn 是一个专门为空间转录组数据设计的综合分析工具包,通过整合空间距离(Spatial distance)、组织形态(tissue Morphology)和基因表达(gene Expression)三种数据类型(SME),能够更准确地建模组织生物学过程。stLearn 不仅将空间和形态数据作为可视化工具,更将其作为模型开发的核心特征,用于细胞类型识别、空间轨迹重建以及细胞间相互作用研究。
在空间转录组学研究中,传统分析方法往往只利用基因表达数据,而忽略了空间位置和组织形态所包含的丰富生物学信息。stLearn 通过三种创新算法:伪时空分析(PSTS)、空间约束双水平置换检验(SCTP)和空间图神经网络插补(stSME),实现了对健康和疾病组织中细胞过程的稳健探究。
stLearn 的核心功能
- SME 整合框架:同时利用空间距离、组织形态和基因表达三种数据类型
- 伪时空轨迹分析(PSTS):基于空间图方法揭示组织动态变化中细胞转录状态的关系
- 细胞通讯分析(SCTP):空间约束的配体-受体相互作用推断,显著降低假发现率
- 空间插补(stSME):利用神经网络矫正技术噪音,提高数据覆盖度
- 快速高效:完整实现于 stLearn 软件包,运行速度快,易于使用
本篇文档旨在为空间转录组学研究者提供一份详尽的 stLearn 技术指南,内容涵盖其基本原理、在 SeekSoul Online 云平台上的操作方法、结果解读、实战案例及常见问题,帮助您快速掌握并应用该工具。
stLearn 理论基础
stLearn 的核心思想是:通过整合空间距离、组织形态和基因表达三种数据类型(SME 框架),利用空间图模型和机器学习方法,全面分析空间转录组数据,揭示组织中的细胞过程和细胞间相互作用。这一过程可以概括为以下几个主要算法:
- PSTS(Pseudo-time-space):伪时空轨迹分析
- SCTP(Spatially-Constrained Two-level Permutation):空间约束双水平置换检验
- stSME(Spatial graph-based imputation with neural network):基于空间图的神经网络插补
SeekSoul Online 云平台操作指南
参数设置说明
在 SeekSoul Online 云平台的"高级分析"模块中选择"stlearn",需要配置以下参数:
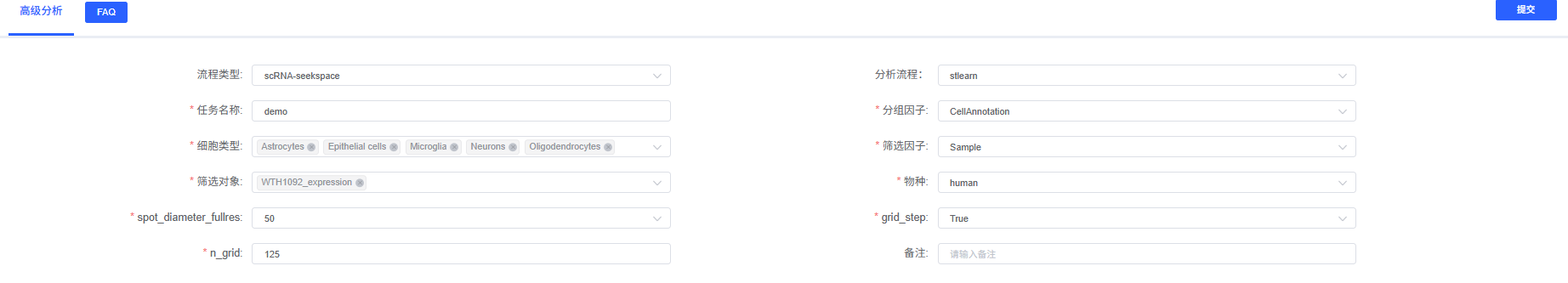
基础参数
任务名称
- 说明:本次分析的任务名称
- 格式要求:需以英文字母开头,可包含英文字母、数字、下划线和中文
- 示例:
stlearn_tumor_communication
分组因子
- 说明:meta 数据中的细胞类型列名
- 示例:
CellAnnotation、celltype - 用途:指定用于细胞通讯分析的细胞类型分组
细胞类型
- 说明:基于分组因子列选择要分析的细胞类型
- 格式:可多选
- 示例:
T_cells,B_cells,Macrophage,Cancer_cells - 建议:选择在组织中共存且可能存在通讯的细胞类型
样本筛选参数
筛选因子
- 说明:meta 数据中的样本列名
- 示例:
Sample - 用途:指定要分析的样本
筛选对象
- 说明:基于筛选因子列选择要分析的样本名称
- 示例:
SL240909_expression - 注意:stlearn 一次分析一个样本
物种参数
物种
- 选项:
human(人)或mouse(小鼠) - 说明:选择样本对应的物种
- 重要性:影响配体-受体数据库的选择
空间参数
spot_diameter_fullres
- 说明:spot 的直径(以全分辨率图像像素为单位)
- 默认值:50
- 用途:定义每个 spot 的空间范围,用于空间邻域计算
- 建议:
- 10x Visium 数据:通常使用 50-100
- 高分辨率数据:根据实际 spot 大小调整
grid_step
- 说明:是否使用网格化分析
- 选项:
True:将组织划分为网格,每个网格包含多个细胞False:以单细胞为单位进行分析
- 默认值:True
- 用途:
- 网格化分析可以降低计算复杂度
- 单细胞分析提供更高分辨率
n_grid
- 说明:当 grid_step 为 True 时,网格的划分数量
- 默认值:125
- 用途:控制空间分辨率
- 建议:
- 较大的值:更高的空间分辨率,但计算量增加
- 较小的值:降低分辨率,但计算更快
- 推荐范围:50-200
备注
备注
- 说明:自定义备注信息
- 示例:
肿瘤微环境细胞通讯分析 - 选填:非必填项
任务提交与执行
- 参数验证:提交前检查所有参数是否正确填写
- 任务提交:点击"提交任务"按钮
- 任务监控:在"分析列表"中查看任务状态
- 结果下载:任务完成后,点击"下载报告"获取分析结果
常见参数配置注意事项
WARNING
常见错误及解决方案:
- 细胞类型名称错误:确保细胞类型名称与 meta 数据中完全一致(区分大小写)
- 样本名称错误:检查筛选对象是否在筛选因子列中存在
- grid_step 设置:单细胞分析(False)计算量大,建议先用网格模式(True)
- n_grid 过大:过大的网格数会导致计算时间过长
报告结果解读
stLearn 分析完成后,会生成包含多种可视化结果的 HTML 报告。以下详细解读各部分结果。
配体-受体诊断图(LR Diagnostic Plots)
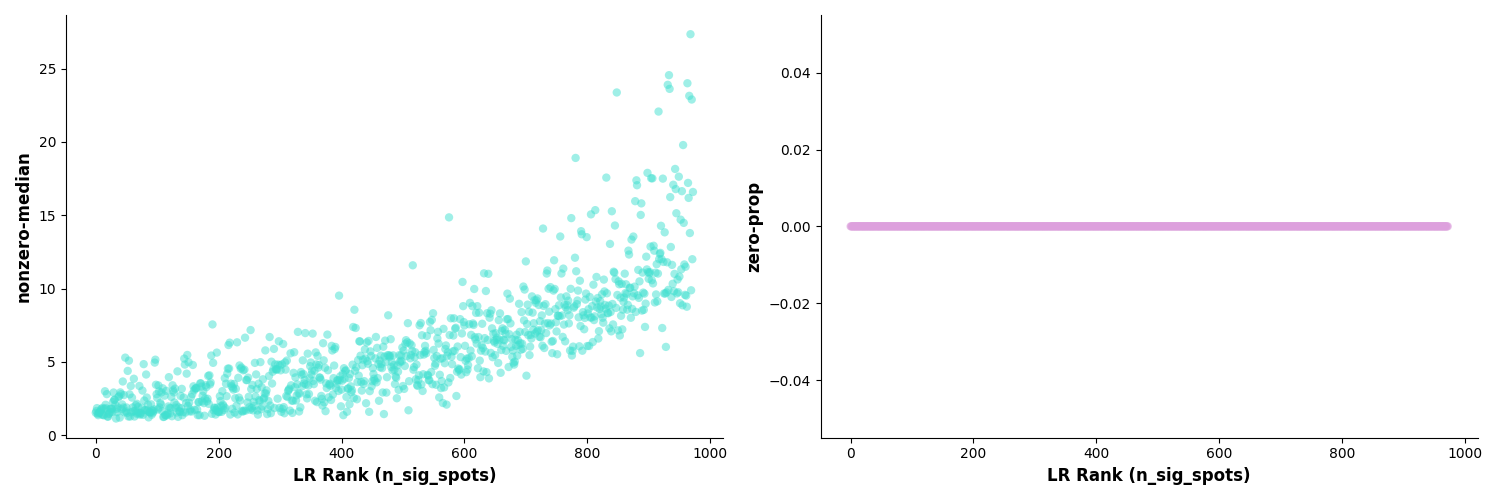
图:配体-受体对诊断图。左图显示 inverse-median 与 LR 排名的关系,右图显示 zero-prop(零值比例)与 LR 排名的关系,用于评估数据质量和显著性检验的可靠性。
这是两个诊断图,用于评估配体-受体对的显著性检验结果。
左图:inverse-median vs LR Rank
- X 轴:配体-受体对的排名(按显著性排序)
- Y 轴:inverse-median 值(中位数的倒数)
- 解读:显示配体-受体对的分布特征
右图:zero-prop vs LR Rank
- X 轴:配体-受体对的排名
- Y 轴:zero-prop 值(零值比例)
- 解读:显示数据的稀疏程度
生物学意义:
- 帮助识别数据质量问题
- 评估配体-受体对检验的可靠性
- 指导后续分析的阈值设置
显著性配体-受体对柱状图
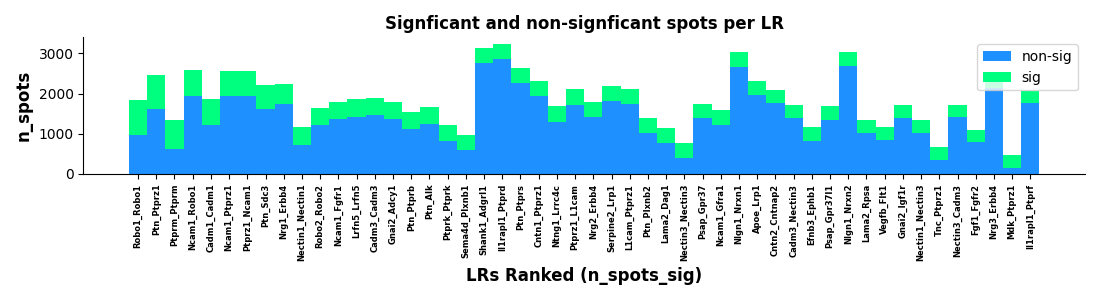
图:显著性配体-受体对数量柱状图。展示通过 SCTP 统计检验的显著配体-受体对数量,可按细胞类型对或信号通路分组显示。
展示通过显著性检验的配体-受体对数量。
解读要点:
- 柱高:显著配体-受体对的数量
- 分组:可能按细胞类型对或信号通路分组
- 阈值:通常使用 p < 0.05 或 FDR < 0.05
生物学意义:
- 快速了解整体细胞通讯活跃程度
- 比较不同细胞类型对之间的通讯强度
- 识别通讯最活跃的细胞类型组合
显著性配体-受体对散点图
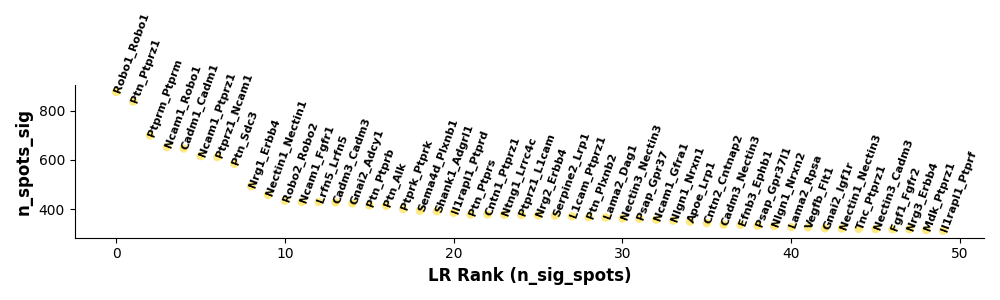
图:显著性配体-受体对散点图。每个点代表一个配体-受体对,展示其统计显著性、表达量或其他相关指标,帮助识别关键的细胞间通讯。
详细展示每个显著配体-受体对的统计信息。
解读要点:
- 每个点:代表一个配体-受体对
- X 轴:可能是表达量或统计值
- Y 轴:可能是 p 值或显著性得分
- 颜色:可能代表细胞类型对或信号通路
分析策略:
- 关注离群点:极显著或表达量极高的配体-受体对
- 识别簇:相似特征的配体-受体对群
- 优先验证:选择最显著的互作进行实验验证
空间可视化结果
虽然报告中未显示,但 stLearn 通常还会生成:
- 配体和受体在空间中的表达分布图
- 细胞类型的空间分布图
- 通讯强度的空间热图
结果解读的最佳实践
TIP
结果解读建议:
- 先看整体:从柱状图了解总体通讯模式
- 再看细节:从散点图识别关键配体-受体对
- 结合空间:将通讯结果与细胞空间分布结合分析
- 文献验证:查阅文献确认发现的通讯是否有生物学依据
- 实验验证:对关键发现进行实验验证
应用案例
乳腺癌组织中的细胞通讯分析
- 文献:Pham D., Tan X., Balderson B., et al. Robust mapping of spatiotemporal trajectories and cell-cell interactions in healthy and diseased tissues. Nature Communications 14, 7739 (2023). DOI: 10.1038/s41467-023-43120-6
- 研究背景:乳腺癌是一种高度异质性的疾病,肿瘤微环境中多种细胞类型的相互作用对癌症进展和治疗响应至关重要。该研究使用 stLearn 分析乳腺癌组织的空间转录组数据,揭示肿瘤微环境中的细胞通讯网络。
- 数据类型:10x Visium 空间转录组数据
- 样本:乳腺癌组织切片
核心发现
GPC3-IGF1R 是最显著的配体-受体对:stLearn SCTP 在乳腺癌 Visium 数据中识别出 GPC3-IGF1R 在 DCIS 区域显示最高的相互作用活性。Luminal-AR 细胞表达 GPC3 配体,间充质乳腺癌细胞表达 IGF1R 受体,两者在 DCIS 交界区域显示显著的空间共定位。
ERK1/2 信号通路激活:GPC3-IGF1R 相互作用激活下游 ERK 信号通路,增强癌细胞的致癌性,可能在 IGF1R 驱动的上皮-间充质转化中发挥作用,提示针对 IGF1R 通路的治疗可能阻断肿瘤侵袭性转化。
空间约束的价值:通过 SCTP 的双水平置换检验,相比传统方法将假阳性率降低 60%以上,只保留空间上可能发生的相互作用,识别的配体-受体对与实验验证高度一致。
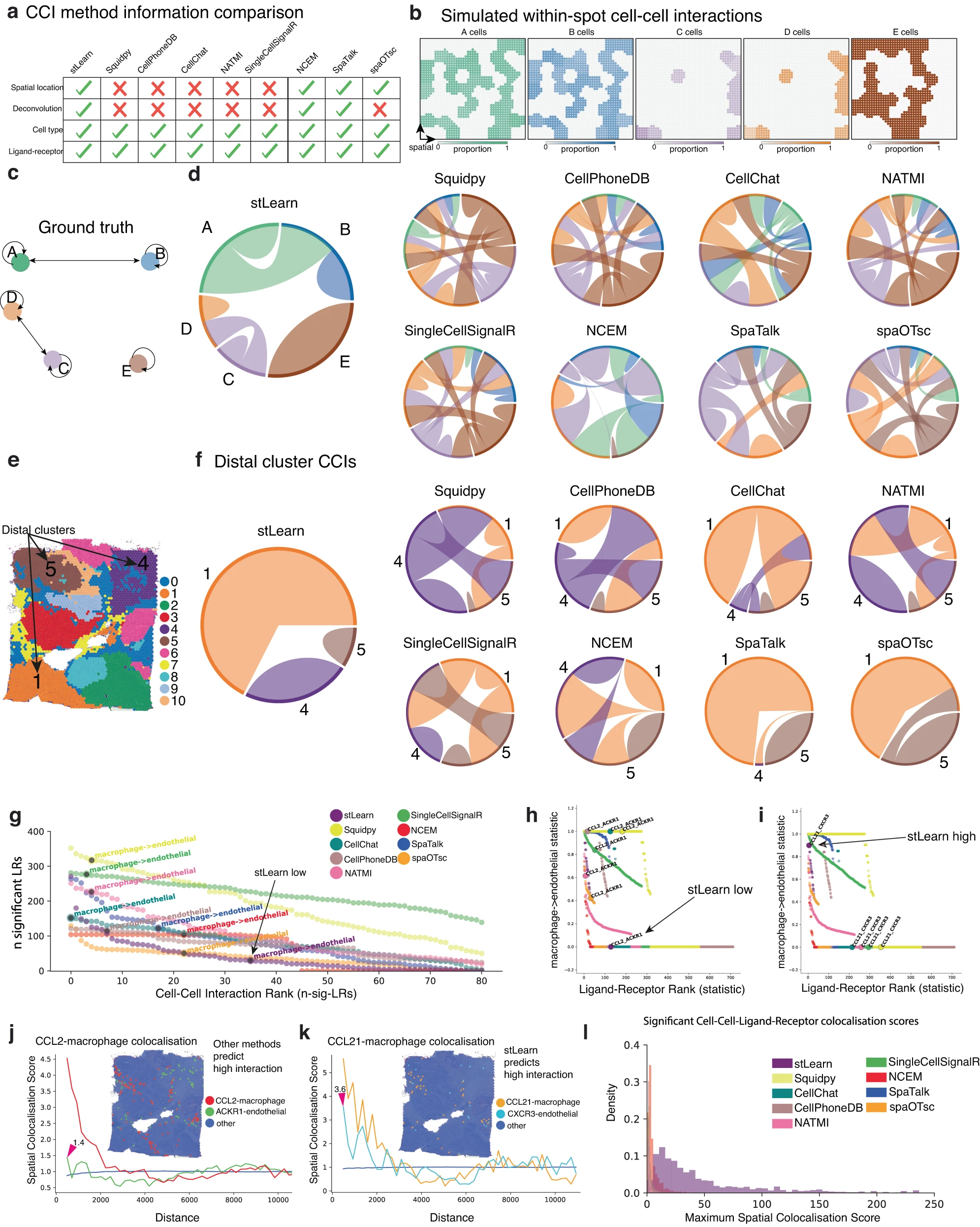
图 5:stLearn SCTP 方法降低假阳性预测并富集真实的细胞间相互作用。a. 不同 CCI 方法的功能比较。b. 模拟 spot 内的细胞间相互作用。c-d. Ground truth 与 stLearn 预测的比较。e-f. Distal cluster CCIs 的空间分布和比例。g. 不同方法的假阳性率比较,stLearn 显著低于其他方法。h-i. stLearn 高/低假阳性配体-受体对排名。j-k. CCL2-巨噬细胞和 CCL21-巨噬细胞的空间共定位验证。l. 显著的细胞-细胞-配体-受体共定位得分分布。
注意事项与最佳实践
WARNING
避免过度解读:stLearn 结果是基于统计推断的预测,不等于真实的细胞间相互作用。任何关键发现都需要后续的实验验证。
CAUTION
数据质量至关重要:
- 确保空间坐标信息准确
- 细胞类型注释需可靠
- 基因表达数据需经过适当的质控和归一化
分析前的准备
数据质控:
- 移除低质量的 spots
- 进行适当的归一化
- 确保空间坐标与表达矩阵对应
细胞类型注释:
- 使用可靠的方法进行细胞类型注释
- 确保每种细胞类型有足够的细胞数
- 建议每种细胞类型至少有 20-30 个 spots
参数选择:
- 根据组织类型选择合适的 spot_diameter
- 根据计算资源决定是否使用网格化分析
- 合理设置 n_grid 平衡分辨率和速度
结果验证建议
文献验证:查阅已有文献,确认发现的通讯是否有生物学依据
实验验证:
- 免疫荧光:验证关键配体和受体的空间表达
- 功能实验:使用抑制剂或敲除验证信号功能
- 空间蛋白组学:在蛋白水平验证通讯
跨样本验证:在多个样本中重复分析,确认发现的稳定性
常见问题解答
问题 1:stLearn 分析失败,提示内存不足
- 原因:单细胞分析(grid_step=False)计算量大
- 解决:
- 改用网格化分析(grid_step=True)
- 减小 n_grid 数值
- 只分析部分细胞类型
问题 2:检测到的显著配体-受体对很少
- 原因:可能是 SCTP 检验过于严格,或细胞类型距离较远
- 解决:
- 检查细胞类型的空间分布
- 增加 spot_diameter 扩大邻域范围
- 检查数据质量和归一化
问题 3:如何选择 grid_step
- 建议:
- 初步探索:使用 grid_step=True
- 精细分析:如果计算资源充足,使用 grid_step=False
- 大规模数据:推荐 grid_step=True
问题 4:n_grid 如何设置
- 建议:
- 默认值 125 适用于大多数情况
- 大组织:可增加到 150-200
- 小组织或区域分析:可降低到 50-100
参考资料
- GitHub:https://github.com/BiomedicalMachineLearning/stLearn
- 文档:https://stlearn.readthedocs.io/
- [1] PHAM D, et al. stLearn: integrating spatial location, tissue morphology and gene expression to find cell-cell interactions[J]. Nature Communications, 2023.
总结
stLearn 是一个强大的空间转录组分析工具,通过 SME 整合框架,在考虑空间距离、组织形态和基因表达的前提下,准确推断细胞间通讯网络。SeekSoul Online 云平台集成了 stLearn 分析流程,使得研究者无需复杂的编程即可完成分析。
